深圳市艾斯基因科技有限公司
4 年
手机商铺
- NaN
- 0
- 0
- 1
- 0
推荐产品
公司新闻/正文
万字综述/IF=33.1 | 表观遗传时钟及DNA甲基化生物标志物在大脑健康和脑部疾病研究中的应用
426 人阅读发布时间:2025-10-15 16:56
衰老在整个生命周期中都会对人脑产生深远影响,目前主要通过认知测试和脑成像技术来监测健康与病理性的大脑衰老过程。越来越多研究证明,利用外周标志物来追踪认知功能、认知老化及神经系统疾病,可为实现这些过程的纵向监测提供一种有价值的微创方法。
2025年6月18日,英国爱丁堡大学Riccardo E. Marioni 教授与牛津大学学者合作在《Nature Reviews Neurology》(IF=33.1)杂志上发表了题为“Epigenetic clocks and DNA methylation biomarkers of brain health and disease”的综述文章,阐述了DNA甲基化生物标志物在监测大脑衰老和分层评估神经系统疾病风险方面的应用潜力以及这些生物标志物与多项认知健康指标(包括认知测试数据、脑部MRI测量值和痴呆症)的关联性。重点探讨了表观遗传时钟——这种可应用于多种组织和器官来评估生物年龄的技术,以及基于血液的表观遗传评分(EpiScores),后者能够直接追踪脑相关表型(如认知功能)和神经系统疾病风险因素(如生活方式行为及炎症相关的蛋白质组学标志物)。

核心要点
-
神经系统功能和疾病的外周标志物可为疾病风险预测提供一种有价值的微创辅助手段;
-
DNA甲基化是一种因组织或细胞类型而异的表观遗传修饰,正日益成为生物标志物开发的热门候选对象;
-
来自血液、大脑等组织的甲基化模式,可构建用于评估实际年龄(称为第一代表观遗传时钟)及其他复杂性状(如认知功能、死亡率或蛋白质水平)的生物标志物;
-
为预测健康寿命、生存寿命和生物衰老速率而构建的第二代和第三代表观遗传时钟,与健康结局的关联比第一代时钟更强,但目前均未显示出与痴呆症的稳健关联;
-
针对其他复杂性状(包括炎症蛋白水平和认知功能)的甲基化生物标志物,在追踪神经退行性病变风险因素方面展现出应用潜力;
-
甲基化生物标志物可能提供一种有效的方法来追踪全生命周期的大脑健康状况。
前言
年龄与衰老是认知能力下降和神经退行性疾病最强的风险因素。然而,相同实际年龄的个体可能表现出不同的生物衰老速率,这种差异可能表现为认知能力下降或神经退行性疾病的诊断。量化实际年龄非常简单,但客观评估衰老速率或确定个体的生物年龄则更具挑战性。
过去十年间,基于DNA甲基化(DNAm)的衰老生物标志物研究兴趣激增。这些化学修饰具有动态性和组织特异性。通过分析每个样本中数千个DNA分子的信息,可确定每个CpG位点甲基化胞嘧啶的百分比(图1)。血液中的DNAm模式不仅与多种哺乳动物的实际年龄相关,也与人类大脑健康指标存在关联。
这篇综述系统介绍了加速表观遗传衰老与表观遗传生物标志物的基本概念,以及构建表观遗传时钟的常用统计方法。首先重点探讨以预测实际年龄为目标构建的第一代时钟——包括基于成人血液与多组织样本、死后脑组织样本及新生儿多组织样本开发的模型。在阐述第一代时钟局限性的基础上,作者进一步聚焦以预测衰老相关结局和生物衰老速率(而非实际年龄)为目标的第二代与第三代关键时钟模型。随后系统梳理这三代时钟与脑老化、认知功能及神经系统疾病之间的关联证据,并补充介绍生活方式因素、蛋白质水平及认知功能的表观遗传预测总结——这些均被认为是神经系统疾病的潜在生物标志。最后,总结了表观遗传生物标志物的整体优势与局限性,并对该领域的未来发展方向与机遇提出展望。
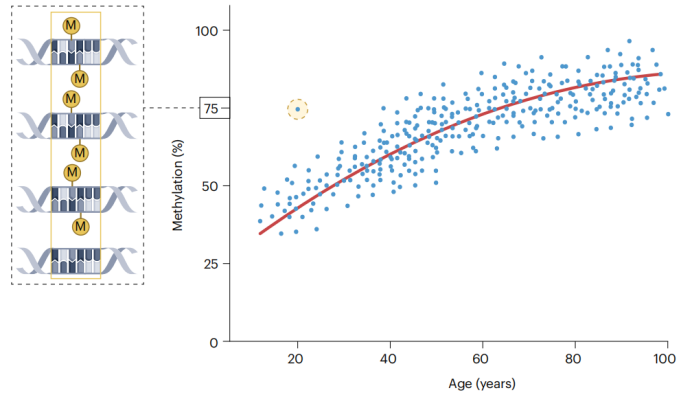 图1 CpG甲基化比例与实际年龄的关系 图1 CpG甲基化比例与实际年龄的关系 |
表观遗传时钟
利用单个CpG位点甲基化比例进行的关联研究为揭示生物衰老的个体差异提供了重要见解。其中重复性最高的衰老相关CpG位点之一是cg16867657,其映射在ELOVL2基因(与脂肪酸延长相关)上,在针对成年生命周期的研究队列中,该位点的甲基化水平与年龄高度相关(Pearson相关系数>0.9)。考虑到这种关联,后续研究的逻辑延伸是通过观测数据与最佳拟合线的偏离程度,来识别个体实际值与年龄预期值的差异,这种偏离可粗略解读为加速或减速衰老。
除单CpG位点与年龄的相关性分析外,研究者常采用弹性网络惩罚回归等统计方法(图2),整合多个年龄相关CpG位点信息构建复合标志物。由此得到的预测值通常被称为表观遗传年龄,其与实际年龄的偏差被定义为表观遗传年龄加速。用于推导表观遗传年龄的加权CpG计算公式可统称为表观遗传时钟。值得注意的是,当在队列中应用表观遗传时钟时,年龄加速偏差与实际年龄之间可能仍存在关联。为消除这一潜在混杂因素,研究者通常采用表观遗传年龄对实际年龄进行线性回归,用获得的残差(即经年龄校正的加速残差)替代原始差异值。
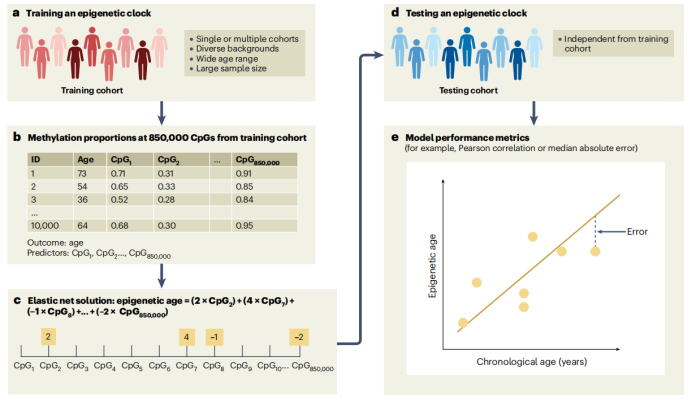 图2 第一代表观遗传时钟的训练和测试 图2 第一代表观遗传时钟的训练和测试 |
表观遗传时钟的构建
在表观遗传时钟的开发过程中,惩罚回归方法可将全部或用户指定范围内的CpG位点子集作为潜在预测特征(称为预测变量或自变量)。尽管全甲基化组测序方法能够捕获基因组中约3000万个CpG位点,但基于芯片的方法在队列研究中得到最广泛验证并应用于表观遗传时钟的开发。图2简要展示了第一代表观遗传时钟的构建流程:首先需要训练数据集(可以是单一队列或联合队列),为确保时钟模型在不同群体中的普适性,理想训练集应包含来自不同背景和覆盖广泛年龄范围个体的均等混合。若使用同质性较高的队列(如单一队列或窄年龄范围),观察到的甲基化模式差异可能无法反映其他群体和年龄段的特征。模型构建后,需在未参与训练的外部测试数据集(称为测试集)中验证性能,常用预测准确性评估指标包括表观遗传年龄与实际年龄的Pearson相关性,以及两者差异的中位数绝对误差或均方根误差。
第一代表观遗传时钟
以实际年龄为预测目标的表观遗传时钟通常称为第一代时钟,而基于衰老相关结局(如全因死亡率)训练的时钟则称为第二代时钟(图3)。最早的第一代表观遗传时钟于2011年被报道,这些时钟包括一个基于68名个体唾液样本训练的预测模型,以及一个基于130份样本(真皮、表皮、宫颈涂片和血液)训练的多细胞/多组织预测模型。这两个时钟均使用Illumina 27K芯片的甲基化数据,在测试集中的平均绝对误差分别约为5年和10年。
2013年1月,该领域迎来了一个阶段性突破:Hannum等人发布了一个基于71个CpG位点的血液时钟模型,该模型基于482名个体训练而成。同年12月,Horvath报道了一个包含353个CpG位点的泛组织时钟,该时钟基于来自50多种组织的约8000份样本的累积数据构建。这两个时钟在测试集中均表现出表观遗传年龄与实际年龄的Pearson相关系数>0.9,预测中位数绝对误差约为4年。这些研究取得了广泛发现,包括描述了表观遗传衰老的性别差异(男性的表观遗传年龄估计值更高)、人类训练的时钟在灵长类动物中的适用性,以及胚胎干细胞的预测年龄接近零。此后,这两种时钟被广泛应用于人类队列研究,其中重复性最高的关联之一是与全因死亡率,即表观遗传年龄加速与寿命缩短有关。
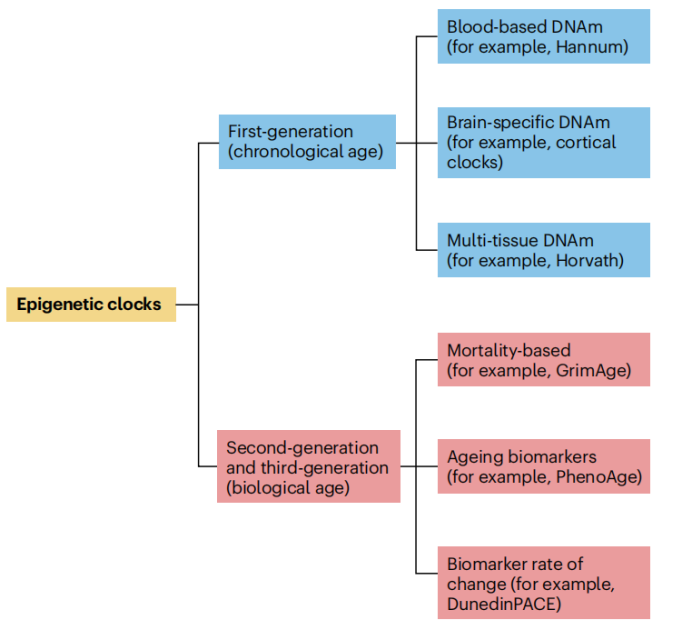 图3 第一代,第二代和第三代表观遗传时钟 图3 第一代,第二代和第三代表观遗传时钟 |
基于大脑的表观遗传时钟
尽管从可及性角度看,基于血液或唾液的预测因子最具优势,将外周组织时钟应用于脑特异性病理研究可能会限制可获得的生物学见解。基于死后皮层组织开发的时钟已在神经退行性疾病患者和健康人群中得到验证,研究发现其与神经退行性临床标志物的关联强度显著优于多组织或血液时钟。图4系统梳理了目前已开发的皮层表观遗传时钟。该领域首.个时钟由Shireby等人通过弹性网络回归开发,使用非阿尔茨海默病个体的死后皮质DNA甲基化数据(样本年龄1-108岁,n=1,397),训练出包含347个CpG位点的DNAmClockCortical模型,该时钟在同期队列的保留样本(n=350)和独立队列的皮质组织样本(n=1,221,年龄41-104岁)中均表现出良好验证效果。值得注意的是,该时钟所选CpG位点与既往血液或多组织时钟的重合度不足5%。在脑组织年龄预测方面,DNAmClockCortical显著优于基于外周组织构建的时钟,但在利用血液DNA甲基化数据预测年龄时表现不及其他多组织或血液时钟。
 图4 皮层时钟发展历程 |
数据集之间的一致性——例如,使用皮层组织同时作为训练数据集和预测样本的基础——是开发和测试表观遗传时钟时需考虑的重要因素。此外,组织的构成也极为关键:例如,由异质性细胞类型组成的混合皮质组织与特定细胞群存在显著差异,因为神经元/非神经元细胞群在神经退行性疾病进展过程中会发生实质性变化。
表观遗传时钟在大脑衰老研究中的应用尚处于起步阶段,特别是在人脑组织样本中,目前仅有少数脑区特异性或组织特异性时钟被开发,例如海马衰老时钟至今仅在小鼠模型中建立。鉴于DNAm模式具有动态性,且时钟主要应用于活体个体,开发更易获取的、基于外周组织替代物的脑衰老预测指标具有充分的合理性。外周生物样本为评估DNAm纵向变化轨迹、以及探索该轨迹是否与脑功能指标或疾病进展指标同步变化提供了常规研究途径。此外,尽管目前尚未出现基于脑脊液(CSF)的DNAm作为训练数据开发出基于脑脊液的时钟,但通过一系列以血液为训练样本的表观遗传时钟估算出的DNA甲基化年龄,已在脑脊液中得到计算。同样,已有多项研究将基于外周血的时钟或泛组织时钟应用于死后脑组织研究。
总体而言,DNA甲基化时钟的性能在老年阶段会进入平台期,导致预测年龄与实际年龄出现偏差。多种表观遗传时钟显示,其预测准确性会随年龄增长呈现非线性变化,且会系统性低估60岁以上人群的预测值。在对皮质组织中多种时钟的检测中,DNAm Clock Cortical时钟表现出最小的年龄相关偏差,这印证了组织特异性时钟的优势。
目前,关于多组织时钟能否准确捕捉不同组织类型衰老速率的问题仍存争议,早期研究提示小脑DNAm衰老速率慢于其他脑区,但新近证据表明当前多组织DNAm时钟会显著低估小脑的表观遗传年龄,尤其当训练数据中脑组织样本占比不足时。在该项研究中,针对752例个体小脑组织的研究鉴定出613个与年龄相关CpG位点,其中仅201个与小脑外皮质区域重叠,这凸显了开发表观遗传时钟时考虑组织特异性甲基化变化的重要性。
总体而言,开发组织特异性时钟不仅对深入理解脑衰老至关重要,同时也亟需更易获取的预测指标,以提示脑健康轨迹的异常偏离。表观遗传时钟可能需要针对特定疾病进程进行定制,并重点关注在大脑与其他组织中高度相关的CpG位点,从而提升其预测准确性。
妊娠期观遗传时钟
鉴于DNAm谱在发育期间会快速变化,研究人员针对这一生命阶段开发了专用时钟,这类时钟旨在精确估计胎龄,有助于识别偏离正常发育轨迹的情况,这可能预示不良健康结局。临床上,精确预测胎龄的需求由来已久,即使仅1周的差异,也可能影响新生儿的发病率、死亡率和出生后的长期健康状况。例如,在关键发育阶段,胎儿若暴露于早产、围产期炎症疾病负担等不同风险因素,其出现神经发育异常及认知功能障碍等不良结局的风险会显著升高。
由于孕期存在独特的共暴露组(指母体与胎儿共同接触的环境、生理等暴露因素),相较于生命其他阶段,选择合适的组织类型对构建精准的妊娠期表观遗传时钟而言,重要性更为突出。目前,妊娠期表观遗传时钟的开发已涉及多种组织,包括脐带血、胎盘组织及颊黏膜细胞;已开发的时钟包括儿童颊黏膜表观遗传时钟(PedBE clock)和新生儿年龄表观遗传估算器,其中PedBE时钟已被证实与不良神经发育结局存在关联。
这些组织的不同特性决定了它们在特定研究方向中的适用性:胎盘作为母体与胎儿系统间的关键媒介,在研究母胎轴相关领域(尤其是胎儿发育表观基因组、妊娠并发症及母体生活方式影响等)中具有极高的相关性 ;而在针对极早产儿(胎龄<32周)的研究中,颊黏膜细胞和唾液常成为首.选样本——因其可通过无创方式采集。此外,胚胎发育阶段,颊黏膜细胞与中枢神经系统(CNS)细胞均起源于外胚层,这一共同起源使其有望为研究神经发育过程提供重要线索;同时,与唾液相比,颊黏膜细胞还能提供更高比例的上皮细胞,且细菌污染程度更低。
局限性与未来优化方向
所有用于预测实足年龄的表观遗传时钟均存在局限性,其中最主要的一点或许是它们无法特异性捕捉疾病风险。尽管实足年龄是多种疾病(包括晚发性神经退行性疾病)最重要的风险因素之一,但表观遗传年龄加速的偏差并非为提供疾病特异性信息而设计。研究表明,随着训练样本量的增加,实现近乎完美的生理年龄预测(或可用于法医学领域)将变得可行,使用较小训练样本构建的时钟往往包含在不同白细胞类型中存在变异的CpG位点,而白细胞比例的差异也与全身性健康状况不佳相关,这可能导致此类时钟与全因死亡率之间出现关联。不过,这些问题会随着训练样本量的扩大而逐渐减少。
一项2023年发表的研究表明,实足年龄预测的中位绝对误差可降至2岁以下。该研究使用的模型,以来自11个队列的24,674份生物样本进行训练,且对CpG位点进行了预筛选,以选取那些与年龄呈现线性或二次关联的位点。考虑到发育阶段观察到的CpG甲基化快速变化及老年阶段出现的甲基化饱和效应,在模型中纳入非线性关联及相应分析方法,或许能为年龄预测研究提供更多新的见解。
第二代与第三代表观遗传时钟
为克服第一代时钟(以实足年龄为训练目标)存在的部分问题,研究人员正以健康相关指标为预测目标开发第二代时钟。目前最广泛应用的第二代时钟包括GrimAge时钟和PhenoAge时钟。
这两种时钟在构建方法上存在细微差异:
-
GrimAge采用两阶段方法:第一阶段通过弹性网络回归生成一系列血浆蛋白和吸烟包年的DNA甲基化特征,随后将这些甲基化替代指标与年龄、性别共同作为输入变量,纳入第二阶段的Cox弹性网络回归中(类似于图2所示模型,但以事件发生时间替代实际年龄),该阶段以全因死亡率为核心结局指标。
-
PhenoAge同样采用两阶段策略:但第一阶段先通过Cox惩罚回归模型,构建全因死亡率的生物标志物及风险因素预测模型,随后使用弹性网络回归(图2)针对该生物标志物的预测模型为目标构建DNA甲基化表观遗传时钟。
另外两种时钟——DunedinPoAm时钟和DunedinPACE时钟,分别用于追踪18种和19种衰老生物标志物的纵向变化速率。前者测量的是26至38岁期间三个时间点的变化率,而后者考察的是26至45岁期间四个时间点的变化轨迹。研究人员通过生成综合变化轨迹,并针对该变化速率训练出表观遗传预测模型。由于这些Dunedin时钟捕捉的是变化速率(而非实际年龄或全因死亡时间这类静态指标),因此被称为第三代时钟。
近年来,研究人员还提出了其他多种表观时钟模型,包括GrimAge时钟的升级版本;用于追踪个体体能差异的FitAge时钟;基于8000多名成年人颊黏膜细胞数据集训练的CheekAge时钟;以及旨在刻画随年龄增长的有害与有益DNAm变化特征的DamAge时钟和AdaptAge时钟。这些时钟模型所使用的CpG位点数量存在显著差异。
第二代表观时钟能否捕捉衰老过程中与健康和疾病相关的不同方面,取决于其预测目标及训练数据集的队列特征。值得注意的是,多种疾病组合可能导致相同的表观遗传年龄估计结果,不同疾病(如癌症、心血管疾病或神经退行性疾病)对个体健康状态的影响仍存在差异。目前,缺乏对生物衰老统一的表型定义,这是表观遗传时钟开发过程中的一个明显局限性。针对性的疾病预测工具或时钟或许有助于突破这一局限,但对于痴呆症等潜伏期长且发病年龄难以精确界定的疾病,相关研究仍面临挑战。即使是对于死因别死亡率这类可精确测量的结局指标也存在困难,因为死亡证明中痴呆症的上报率通常较低。
表观遗传时钟与认知功能、痴呆症及生活方式因素的关联性
鉴于表观遗传时钟种类繁多,且认知功能与脑健康结局指标的衡量维度存在差异,综合分析两者间的关联数据具有一定的复杂性。现有的研究样本量通常仅为数千例,在整合结果时会遇到显著的异质性。尽管如此,已有学者开展了系统性综述。在痴呆症、轻度认知障碍和认知功能方面:Zhou等学者整合了截至2022年发表时的30项研究证据,涵盖Horvath、Hannum、GrimAge、PhenoAge和DunedinPACE等主流时钟,针对包含多种神经退行性疾病的痴呆症表型,样本量普遍偏小(每项研究n<1,000),表观年龄加速与认知结局存在强关联的证据仍然不足。McCrory等于2021年发表的一项研究(未纳入前述系统评价)得出了类似结论,该研究利用爱尔兰老龄化纵向研究(TILDA)490名参与者的数据发现,基于Hannum和Horvath时钟的表观年龄加速与认知功能及其他健康指标均无显著关联。相比之下,在调整少量混杂因素的模型中,PhenoAge时钟和GrimAge时钟的衰老加速与整体认知评分存在相关性,但与反应时指标无关,当纳入更严格的协变量后,GrimAge加速的显著关联仍然存在。另一项研究得出类似结果,PhenoAge、GrimAge和DunedinPACE均与较低认知测试得分存在横断面关联。Chervova等2024年的系统评价指出,表观遗传衰老与认知功能的关联存在不一致性:GrimAge和PhenoAge时钟的衰老加速与认知能力下降相关,然而该研究的荟萃分析显示GrimAge加速与痴呆症无显著关联。
为探究时钟与脑部MRI结局的关联,Whitman等整合了三个数据集的数据,共包含2,322名受试者的3,380次观测数据,这些数据覆盖中年至老年阶段。研究重点分析了五种表观遗传时钟(DunedinPACE时钟、Horvath时钟、Hannum时钟、PhenoAge时钟及 GrimAge时钟)与基于MRI的脑部结构指标之间的关联。结果显示:DunedinPACE时钟和GrimAge时钟的衰老加速,与全脑体积降低、海马体体积降低、白质病变体积增大及皮质变薄的关联最为稳定一致。
在Chervova等人发表综述与荟萃分析之前,Oblak等人已针对表观遗传时钟与各类生物、社会及环境因素的关联,开展了一项全面的系统性综述。该综述纳入了156项研究的结果,涵盖数十种表型关联,并涉及34种潜在的表观遗传时钟——其中28种为预测实足年龄的第一代时钟。在所有时钟中,Hannum时钟、Horvath时钟、PhenoAge时钟及GrimAge时钟的应用最为频繁。在荟萃分析包含的57种因素中,有36种与表观遗传时钟呈现出统计学意义上的显著关联。其中部分因素是已知的脑健康风险因素或相关因素:例如,第一代Hannum时钟和Horvath时钟的衰老加速,与酒精摄入量更高及男性性别相关;Horvath时钟的衰老加速还与体重指数(BMI)升高有关。第二代PhenoAge时钟和GrimAge时钟的衰老加速,则与教育水平较低、体力活动减少及吸烟相关。值得注意的是,GrimAge时钟与吸烟的关联并不意外——因为在该时钟的构建过程中,已纳入了基于甲基化的吸烟替代指标。
第一代与第二代时钟的核心差异之一在于:第二代时钟与精神疾病特征存在关联。具体而言,基于DNAm的PhenoAge时钟和GrimAge时钟均与精神分裂症相关,且GrimAge时钟还与抑郁症相关。总体来看,GrimAge时钟与各因素的关联效应量大于其他时钟。
尽管已有研究普遍显示,基于血液的DNAm年龄(或衰老加速)与认知能力指标存在相关趋势,但鲜有研究能够同时探讨神经病理学特征对这一关联的混杂影响。不过,有一项研究对宗教团体研究和拉什记忆与衰老项目(合称 ROSMAP)参与者的死后背外侧前额叶皮质组织样本,计算Horvath时钟和 PhenoAge时钟的表观遗传年龄。结果发现,DNAm年龄与认知能力(整体认知及情景记忆)之间约50%的关联,可由神经病理学特征(包括淀粉样蛋白病理改变)的差异来解释。
Beyond clocks:生活方式危险因素的表观遗传生物标志物
除表观遗传时钟与认知健康或神经退行性疾病结局存在直接关联外,已有研究关注了时钟与影响生命全程大脑健康的风险因素之间的关联。如2024年《柳叶刀》痴呆症委员会报告所强调,肥胖、饮酒、吸烟、高血压及高LDL胆固醇水平等众多明确且可干预的风险因素,均会对晚年大脑健康产生影响。
表观基因组关联研究(EWAS)与全基因组关联研究已发现,数千个CpG位点与这些可干预风险因素存在关联。类似表观遗传时钟的开发思路,研究人员也构建了多CpG位点特征标记,为特定形状与行为提供替代测量指标。这种方法对吸烟、饮酒及体重指数等指标具有重要价值。关键的是,研究表明这些基于甲基化的预测因子(有时称为EpiScores或甲基化风险评分)在与健康及疾病相关指标关联分析时,往往能产生更大的效应量。
血浆蛋白的表观遗传生物标志物
GrimAge时钟的构建过程中纳入血浆蛋白的EpiScores,这一设计提示了健康衰老、甲基化组与蛋白质组之间存在复杂相互作用。近年来,蛋白质组在疾病生物标志物的开发领域的关注度不断提升,痴呆症的蛋白质生物标志物研究已初见成效。
英国生物样本库研究证实,如胶质纤维酸性蛋白(GFAP)、潜伏转化生长因子β结合蛋白2(LTBP2)和神经丝轻链(NfL)等单一蛋白及复合蛋白质评分均与全因痴呆风险存在关联。值得注意的是,英国生物样本库数据显示,在痴呆诊断确立前长达15年,血浆GFAP与NfL水平就已出现升高。蛋白质水平本身作为生物标志物潜力巨大,而蛋白质的DNA甲基化替代指标同样能反映与健康结局的类似关联。但现有研究表明,并非所有蛋白都存在对应的替代指标。随着更大规模队列研究开始同步采集DNA 甲基化数据与血浆蛋白质组数据,未来需重点明确两类场景:一是EpiScore的构建具备可行性的场景;二是在预测认知或脑健康结局时,这类评分能够补充甚至优于直接测量的蛋白质特征标记的场景。
此外,越来越多的证据表明,蛋白质EpiScore作为炎症标志物具有特殊价值。慢性低度炎症(即炎性衰老,inflammaging)对健康的负面影响,在神经系统疾病与非神经系统疾病中均日益得到认可,而C反应蛋白(CRP)已被视为反映这一过程的潜在生物标志物。然而,血浆CRP水平对感染、损伤等急性事件高度敏感,这一特性导致单时间点CRP测量值作为长期炎症标志物的可靠性大幅降低。
EpiScores或有助于缓解CRP水平短期波动带来的影响。有多项研究采用这一思路:首先从CRP表观基因组关联研究(EWAS)中获取回归权重,随后在独立样本中构建CRP表观遗传评分,并验证其与多种健康结局的关联。这类表观遗传评分的表现有时优于基于血浆或血清CR水平的指标:在老年人中,它与认知功能及脑部MRI指标等性状的相关性更强。而在生命早期,出生时较高CRP表观遗传评分,与7岁时较差的整体认知功能相关,同时,相较于足月新生儿,早产儿出生时的CRP表观遗传评分还与围产期炎症疾病负担程度、不良脑宏观结构结局(如脑白质、深部灰质、海马体及杏仁核体积减小,以及多条通路的脑白质微结构特征异常)存在关联。另有研究对1000余名8-19岁儿童的唾液DNA甲基化数据进行分析,发现CRP表观遗传评分与认知速度、执行功能、推理能力及语言理解能力的下降相关。
甲基化替代指标法的应用已拓展至其他蛋白质及健康结局研究领域。例如,Gadd等人构建了109种蛋白质的EpiScores,研究发现这些评分与137种新发疾病存在关联,其中包括中风 。在一项涵盖三个苏格兰队列的荟萃分析中,这109种评分中的18种还与整体认知功能相关,但它们与新发痴呆症的关联强度相对较弱。
认知功能的表观遗传生物标志物
目前仅有少数大规模认知功能表观基因组研究得以开展。一项涵盖11个队列(n=2,557-6,809)的荟萃分析,以及两项使用苏格兰世代队列数据的研究(样本量分别为9,162与18,264),仅在成年群体中识别出少量候选位点。其中第二项研究的先导位点与荟萃分析结果及阿尔茨海默病EWAS研究均无重叠。这种差异可能在在于三项研究的分析方法不同。
与单个CpG位点与认知功能关联的研究结果形成对比的是,两项苏格兰世代研究均进行了方差成分分析显示,EPIC芯片上所有CpG位点的甲基化信息,最多可解释词汇测试结果73%的变异。这表明基于血液DNA甲基化数据进行分析,认知功能很可能是一种高度多表观遗传位点调控性状,即由大量微效CpG位点共同作用决定。在外部验证队列中,基于9,162名苏格兰成人训练的认知EpiScore在应用于一项混血儿童及青少年队列时,可解释整体认知功能3.4%的变异;而采用多元回归框架,基于18264名参与者训练的认知EpiScore,对整体认知功能变异的解释度则超过6%。基于9162名苏格兰成年人数据训练的认知EpiScore,还被应用于美国得克萨斯州一个多种族裔儿童青少年队列。结果显示,该评分可解释该队列儿童青少年数学测试成绩个体差异的11%以上。此外,与 该评分与实测认知功能的关联强度 相比,其与脑部影像学指标、生活方式、整体健康状况及蛋白质水平的关联虽方向一致,但强度较弱 。
研究人员还采用机器学习技术,基于痴呆症风险因素与认知功能,从全DNAm数据中构建出多变量甲基化风险评分,用于预测认知障碍的发生。结果表明,相较于CSF生物标志物或多基因风险评分,这类甲基化风险评分能显著提升轻度认知障碍的预测效果——这一发现证实,将表观遗传评分纳入现有风险预测策略具有潜在价值。
与成人领域的研究现状类似,针对新生儿、婴儿及青少年认知功能的EWAS,目前仍以小规模研究为主 。纵观全生命周期,不仅认知功能的小规模EWAS研究较为有限,针对DNA甲基化与脑部MRI所测神经相关性状直接关联的研究同样关注不足。目前规模最大的相关研究之一,是由ENIGMA表观遗传学工作组联盟开展的项目:该研究涵盖11个队列(n=3337),针对丘脑、伏隔核、海马体这三个皮质下结构的体积进行EWAS分析 ,结果仅在海马体体积相关分析中识别出两个CpG位点:cg26927218和cg17858098;在其他结构体积的主要EWAS分析中,未发现其他显著相关的CpG位点。另有一项规模更大的研究,针对14个社区居住成人队列(n=9732)的脑白质高信号体积开展分析,最终识别出12个全表观基因组显著相关的CpG位点,以及46个基于基因组区域的DNA甲基化关联 。
总体而言,目前关于血液甲基化组与认知功能或脑结构指标关联的研究仍相对稀缺。
局限性与未来方向
这篇综述聚焦于CpG位点的DNAm特征,但其他表观遗传特征与修饰(如羟甲基化、组蛋白修饰及染色质可及性变化)也可能呈现出与衰老及大脑相关的模式。然而目前,基于芯片的CpG甲基化检测仍是在大规模、多样化人群队列中应用最广泛、特征最明确的表观遗传修饰。2024年,牛津纳米孔公司宣布计划对英国生物样本库5万份样本进行测序,以构建人类基因组的表观遗传图谱。该测序技术将允许研究人员能够检测约3,000万个CpG甲基化位(相比之下,Illumina芯片仅覆盖约100万个位点),并可同步检测羟甲基化。未来,表观遗传生物标志物研究可进一步拓展:从批量组织(如全血)样本的研究结果,向单细胞数据的转化应用延伸。
将DNA甲基化生物标志物推广至不同人群,并将其转化为临床实践,目前仍面临多项技术、实践及伦理层面的挑战:
技术平台差异:不同检测平台得出的DNA甲基化测量结果可能存在差异,且表观遗传时钟的估算值波动可能很快;
队列普适性与可靠性:以DunedinPACE时钟为例,其训练过程仅采用了约81,000个CpG位点——这些位点同时存在于450K芯片和EPIC芯片中,且具有良好的重测信度;
表观遗传漂变的影响:表观遗传漂变(即随衰老发生的随机性DNA甲基化变化)可能为时钟预测引入噪声,在老年人群中尤为明显,进而影响生物标志物的稳定性;
量化方式的局限性:芯片检测仅能提供甲基化的相对定量结果(而非绝对定量),这为在个体水平解读数据或将个体的表观遗传评分/时钟估算值与参考人群对比带来了挑战;
人群多样性不足:现有研究(尤其是生物标志物训练相关研究)大多聚焦于欧洲裔人群,因此还需进一步研究以明确表观遗传评分与表观遗传时钟在不同人群中的适用性。
尽管表观遗传时钟在捕捉全身性衰老特征方面具有实用价值,但其设计本身缺乏疾病特异性。随着该领域的不断发展,以及更大规模生物银行队列生成更多DNAm数据,未来有望构建一组针对特定疾病或性状的定制化DNAm预测工具。这种方法需依赖统一的疾病结局定义,以及足够数量的病例用于模型训练与验证——相较于第二代、第三代表观遗传时钟(这类时钟基于多种衰老标志物混合训练而成),定制化预测工具可能产生更具解释性和可操作性的结果。
另一项优先研究方向是:探究表观遗传时钟与EpiScores,在已确立的神经退行性疾病生物标志物(如淀粉样蛋白成像、tau 蛋白成像或体液生物标志物)基础上,能否提供额外的预测价值。对这些指标(包括现有生物标志物与表观遗传标志物)进行对比分析,若条件允许,再结合脑特异性表观遗传改变进行研究,将有助于明确基于血液的表观遗传生物标志物在哪些场景下最具应用价值。
若想将表观遗传时钟(无论是疾病特异性时钟,还是通用性衰老标志物)转化至临床场景,克服前文提及的各类挑战至关重要。我们推测,表观遗传生物标志物最有可能的临床应用场景是疾病风险分层——在此场景中,它们可作为现有工具的补充,助力疾病预防与风险降低工作的开展。
结论
为追踪实际年龄与生物衰老的不同维度,目前已开发并持续完善了多种表观遗传时钟。尽管部分时钟的加速速率与特定健康结局存在关联,但目前这类关联性尚未能稳定延伸至痴呆症领域。重要的是,在疾病发病机制中,当前的表观遗传时钟更可能作为生物标志物而非致病因素。此外,相较于使用反映神经退行性病变分子机制的样本(如脑组织),利用外周生物样本(如血液、唾液)训练时钟或许具有更重要的实际意义——因外周样本更易获取,更便于大规模研究与临床应用。此外,针对认知功能、生活方式以及代表神经退行性病变风险因素的蛋白质指标所开发的EpiScores,其应用也为追踪全生命周期脑健康状况提供了一种有效的研究思路与工具。
艾斯基因
深圳市艾斯基因科技有限公司创建于2016年,隶属于国家高新技术企业和专精特新创新型中小企业。核心团队是国内最早一批表观多组学科研技术开拓者之一,在Nature、Cell等及其子刊发表论文数十篇。产品覆盖DNA修饰、RNA修饰、组蛋白修饰/开放染色质等多维度解决方案。同时,提供基因组、转录组、蛋白组、代谢组、暴露组等多组学产品服务,包括实验设计—样本处理—生信分析—数据应用全链条完整解决方案,帮您高效避坑,突破难点!