深圳市艾斯基因科技有限公司
4 年
手机商铺
- NaN
- 0
- 1
- 0
- 3
推荐产品
公司新闻/正文
CUT&Tag:打开表观遗传奥秘的钥匙,这些要点你必须知道!
420 人阅读发布时间:2025-07-08 16:35
背景知识
2019 年 Henikoff 实验室在 Nature Communications 杂志上发表了题为 “CUT&Tag for efficient epigenomicprofiling of small samples and single cells" 的论文,首次系统地提出了 CUT&Tag(Cleavage Under Targets and Tagmentation) 这一新型的表观遗传学分析技术用于蛋白质-DNA相互作用的研究。

技术原理
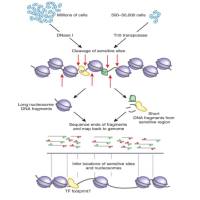
CUT&Tag是一种创新高效的、适合微量样本的DNA-蛋白互作研究技术,主要用于研究转录因子或组蛋白修饰在全基因组上的结合位点或分布特点。CUT&Tag利用Tn5转座酶与Protein A/G的融合蛋白,通过二抗把Tn5酶复合体引导至目标染色质区域同时连接测序接头,直接PCR扩增测序。
CUT&Tag核心技术点在于:
(1)利用抗体把目标蛋白和基因工程改造过的 Tn5 转座酶吸附在一起;
(2)利用 Tn5 酶的转座功能对目标蛋白吸附区附近的 DNA进行切断,并在切下的片段两端加上标签序列,然后构建成深度测序文库;
(3)再通过深度测序和生物信息学分析得到相关信息
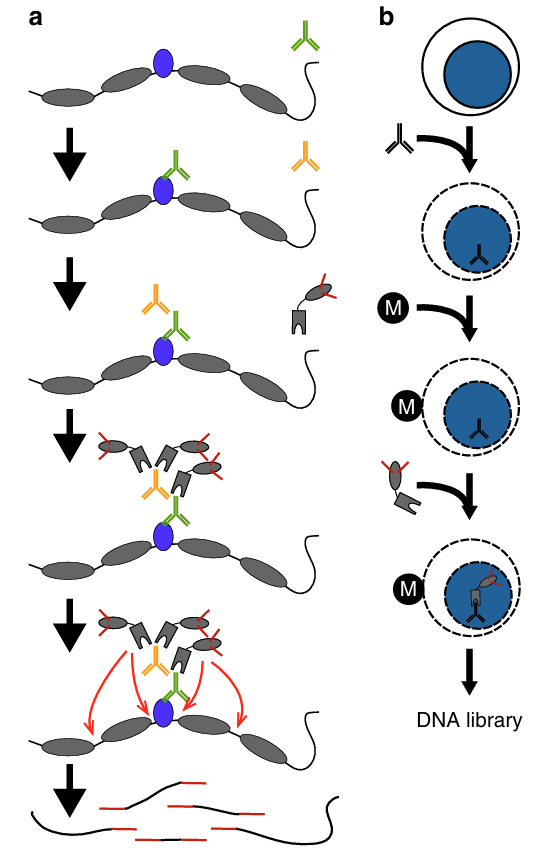
图1 CUT&Tag技术原理
具体步骤:
1. 细胞与ConA 磁珠结合:
使用细胞核提取试剂盒提取细胞核并进行计数。细胞核被分离出来,与预先用刀豆蛋白A(ConA)包被的磁珠混合。ConA能够特异性地结合细胞膜上的糖蛋白,从而将细胞核吸附到磁珠上。
2. —抗孵育:
利用洋地黄皂苷(Digitonin)进行细胞膜通透,使抗体能够穿过细胞膜和核膜进入细胞核识别靶蛋白。将特异性一抗(针对目标蛋白的抗体)加入到细胞核与磁珠的混合物中,在4°C下孵育过夜,使抗体能够特异性地结合到目标蛋白上。
3. 二抗结合:
添加与一抗特异性结合的二抗,在室温下孵育一段时间。不仅可以充当桥接抗体并且可以增加蛋白A/G 结合位点的数量,以增加和放大信号
4. Protein A/G-Tn5转座酶酶切:
Protein A/G-Tn5融合蛋白与抗体复合物结合,将Tn5转座酶精准定位到目标DNA区域。
5. 片段标签化并提取DNA:
pAG-Tn5转座体在添加Mg2+ 离子的高盐洋地黄皂苷缓冲液中,并在37℃下孵育,激活Tn5活性,打断DNA并加上接头序列。使用蛋白酶K消化目的蛋白和抗体,然后使用试剂盒提取基因组DNA;
6. 文库扩增和文库纯化:
对DNA片段进行扩增,再使用合适的DNA纯化磁珠等,将片段化产物中的DNA全部纯化出来,从而获得完整的Cut&Tag测序文库以用于测序。
7. 高通量测序:
使用Illumina等高通量测序技术对文库进行测序。通过测序结果,得到大量与目标蛋白结合的DNA片段序列。
8. 数据分析:
将测序得到的DNA序列比对到参考基因组,确定这些片段在基因组中的具体位置。根据这些位置,绘制目标蛋白的基因组结合图谱,分析它的结合模式和潜在的功能。
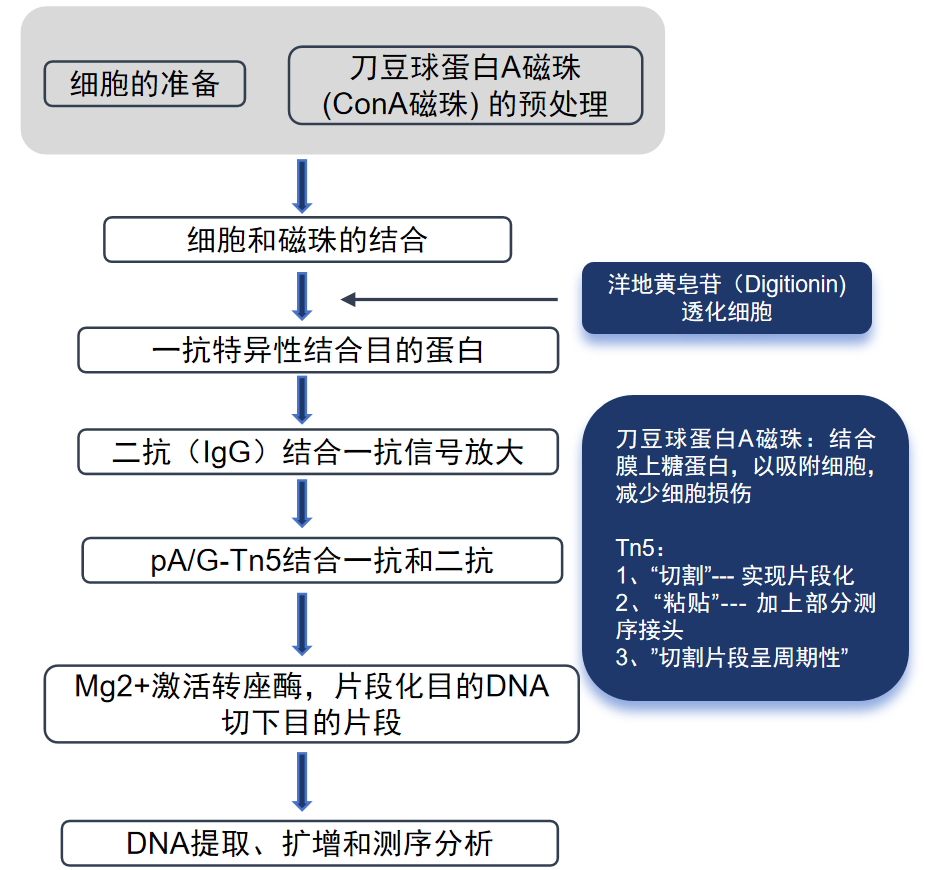
图2 CUT&Tag实验流程
质控标准
-
细胞核质控:
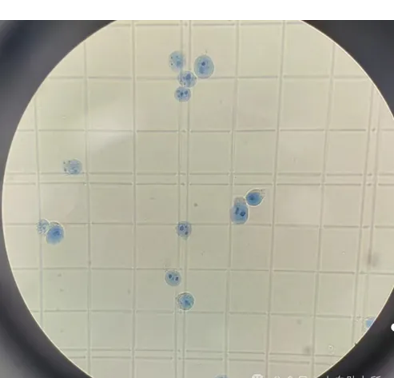
-
文库片段分布质控: 切割片段应该来自于无核小体区域、单核小体区域、双核小体区域、三核小体区域等,在100-200、300-400bp、500-600bp左右位置有明显峰(如图4所示)。而一个失败的文库则可能表现出很均匀的片段分布(如图5所示)。
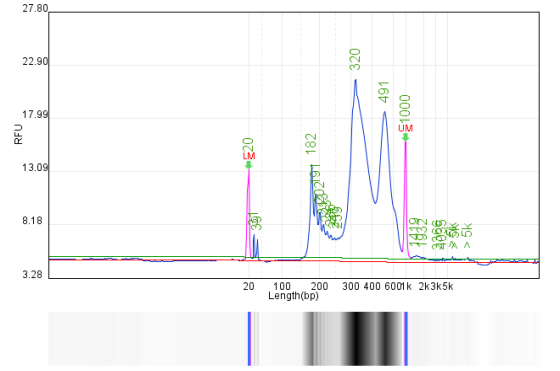
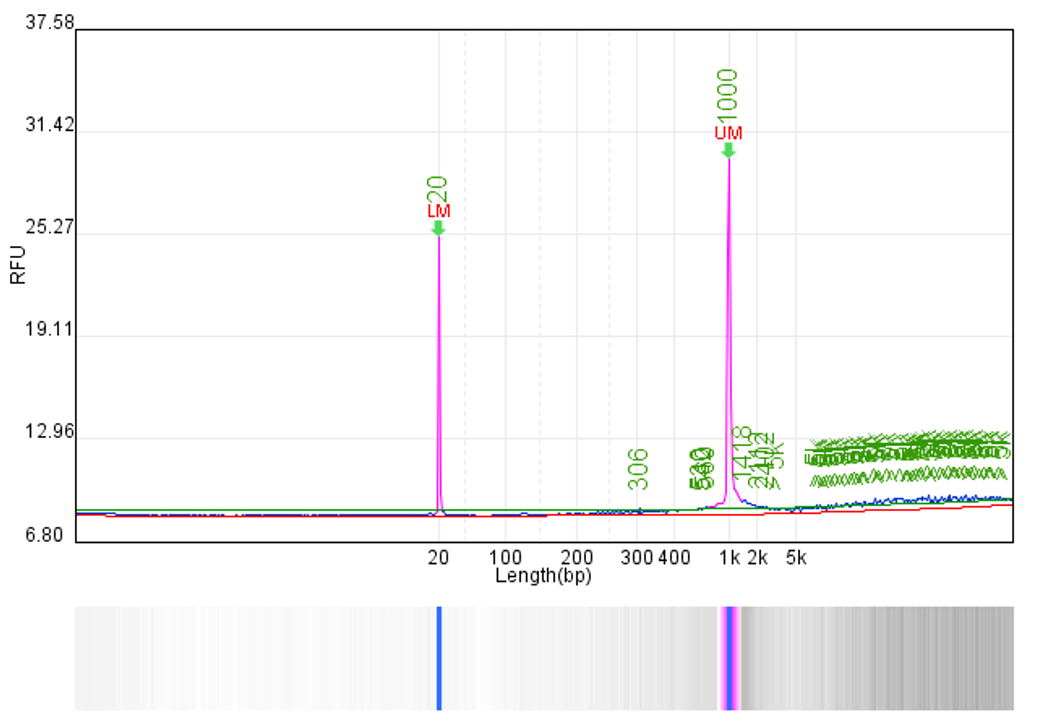
-
数据质控:Mapped rate>60%,Frip>10%
分析内容
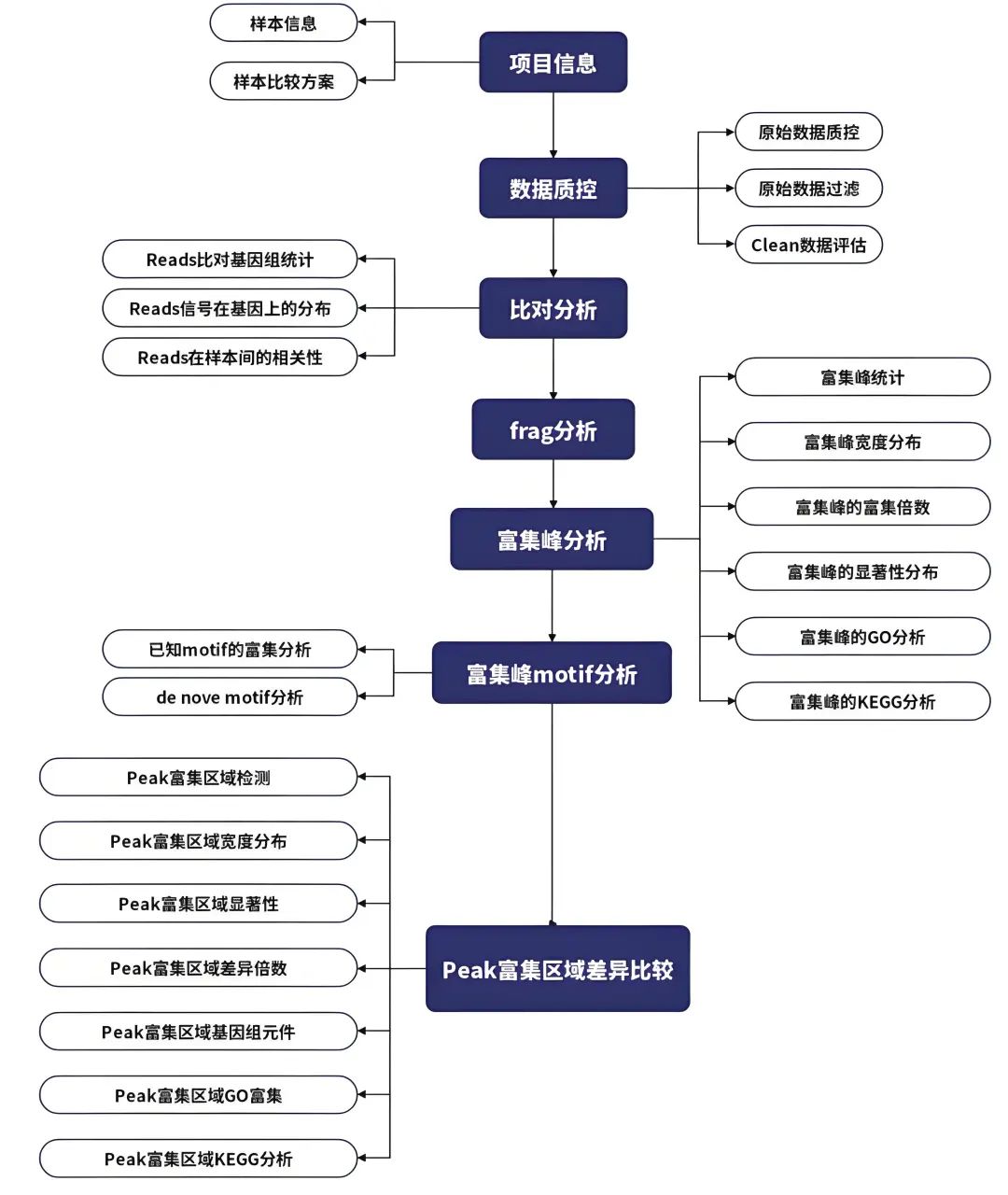
结合学科前沿和研究需求,艾斯基因提供丰富美观的生信报告,多维度展示项目信息。提供数据质控→比对分析→frag分析→富集峰分析→富集峰motif分析→peak富集区域差异比较→个性化分析的深层次数据解析。
技术优势

送样要求