深圳市艾斯基因科技有限公司
4 年
手机商铺
- NaN
- 0
- 1
- 0
- 3
推荐产品
公司新闻/正文
PNAS | 基于深度学习鉴定cfDNA甲基化的组织特异性图谱及定量方法
1320 人阅读发布时间:2023-11-22 18:02
近日,加州大学洛杉矶分校研究团队与斯坦福大学团队、EarlyDiagnosticInc公司团队等合作在PNAS发表了题为“Comprehensive tissue deconvolution of cell-free DNA by deep learning for disease diagnosis and monitoring”的文章。研究中准确鉴定出每个组织特有的DNA甲基化模式,有助于识别与检测发现的cfDNA改变相关的特定组织或器官,同时这也是进行疾病准确诊断和监测所面临的一个关键挑战。
研究摘要
英文标题:Comprehensive tissue deconvolution of cell-free DNA by deep learning for disease diagnosis and monitoring
中文标题:基于深度学习cfDNA 的全面组织反褶积对疾病进行诊断和监测
发表时间:2023年07月11日
发表期刊:PNAS(IF = 11.1 / SCI 一区)
技术方案:RRBS、WGBS、ChIP-seq、cfMethyl-Seq等
样本类型:癌组织+癌旁组织+血浆cfDNA
研究内容
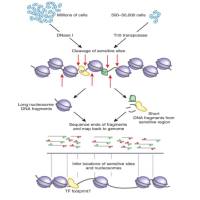
(1)构建全面的组织甲基化图谱研究人员纳入了来自基因型-组织表达(GTEx)项目的521个非癌症个体组织样本的甲基化数据(RRBS),涵盖了29种主要的人体组织。基于这些RRBS数据,研究人员在DNA片段水平上系统地发现了组织特异性甲基化标记物。
(2)cfSort模型介绍 研究人员使用521个组织样本的RRBS数据,以预定义的组织组成和不同覆盖深度生成cfDNA甲基化数据集作为训练、验证和测试数据。研究共生成295484个不同的训练样本和数千个验证和测试样本,满足DNN训练和评估的要求。此外,在数据生成中,研究人员充分利用不同组织类型、不同样本、不同组织组成的组合,探索数据中可能存在的噪声和偏差。因此,cfSort将从这些数据中学习稳健的组织特异性。
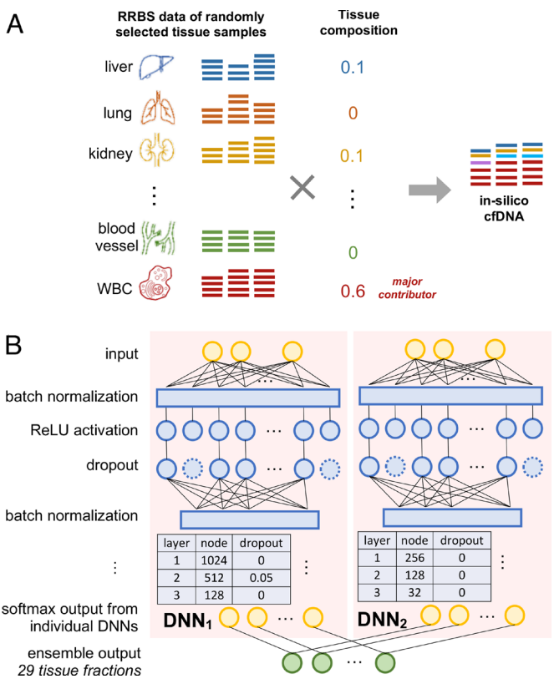
图2 cfSort分析流程图
(3)cfSort模型的性能评估 研究团队在一个独立的测试集上评估了cfSort的性能,将其性能与两种现有的组织反褶积方法进行了比较:非负最小二乘法(NNLS)和CelFiE。使用平均绝对误差、估计的组织分数与真相之间的相关性来评估方法的准确性。cfSort在所有指标上都优于NNLS和CelFiE,表明cfSort在估算组织成分方面的准确性高于其他两种方法。反褶积方法需要有较高的检测限,以低比例检测组织衍生的cfDNA。cfSort能够以0.1%的组织分数检测组织衍生的cfDNA,而NNLS以5%,CelFiE以0.5%检测组织衍生的cfDNA。以上结果表明cfSort比两种竞争方法具有更好的检测下限。
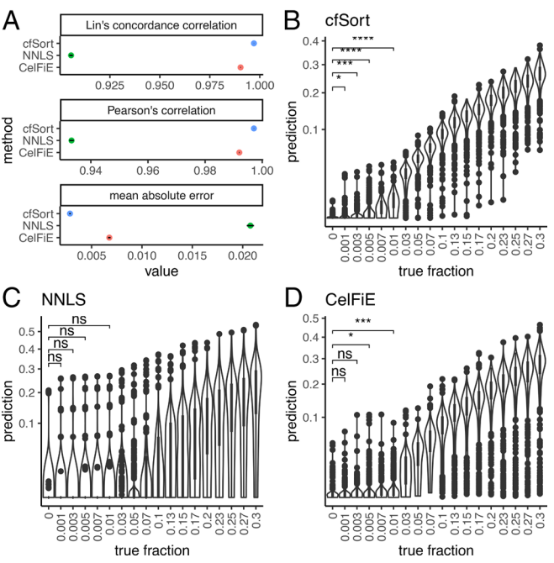 图3 cfSort分析性能的评估与比较
图3 cfSort分析性能的评估与比较
研究小结
综上所述,该研究报告了一个全面的高分辨率组织甲基化图谱和基于cfDNA的监督型组织反卷积方法—cfSort,其能够对cfDNA中的组织部分进行敏感而稳健的定量。研究人员还通过多个独立的数据集验证了组织标记图谱和cfSort的性能,表明这些标记与组织发育、组织分化和组织特异性转录相关。组织甲基化图谱和cfSort增强了cfDNA中组织去卷积的性能,从而促进了基于cfDNA的疾病检测和纵向治疗监测。
参考文献:Li S, et al., Comprehensive tissue deconvolution of cell-free DNA by deep learning for disease diagnosis and monitoring. Proc Natl Acad Sci U S A. 2023 Jul 11;120(28):e2305236120.
原文链接:
https://pubmed.ncbi.nlm.nih.gov/37399400/